- 结果文件
- 使用指南
- 版本记录
- 联系客服
处理和分析微生物数据,采用LEfSe(Linear Discriminant Analysis Effect Size)方法进行特征选择和差异分析,建议每个分组生物学重复数≥5。
LEfSe分析是一种用于发现和解释高维度数据生物标识(基因、通路和分类单元等)的分析工具,可以进行两个或多个分组的比较,它强调统计意义和生物相关性,能够在组与组之间寻找具有统计学差异的生物标识(Biomarker )。该分析首先使用非参数Kruskal-Wallis 秩和检测不同分组间丰度差异显著的物种,然后使用Wilcoxon秩和检验上一步的差异物种在不同组间子分组中的差异一致性,最后采用线性回归分析(LDA)来估算每个组分(物种)丰度对差异效果影响的大小。
1、当所有分组或某一分组未筛选到满足条件特征(显著水平 和 LDA值的阈值)时,小工具可能会运行失败或绘图结果中缺失对应组别结果;
2、绘图结果中的特征数过多或过少时,可通过调整 显著水平(alpha值)和 LDA值的阈值 来优化绘图使用特征数量;
3、绘图结果出现压缩或显示不完整时,可通过调整 LDA分布柱状图高度/宽度 和 物种进化分支图高度/宽度 来改善。
示例文件:
分组信息,第一列为样本ID,第二列为分组(Group),确保列名与示例相一致,支持 .tsv、.txt 和 .csv 格式的表格文件
OTU/特征表,每一列代表一个样本,每一行代表一个特征,支持 .tsv、.txt 和 .csv 格式的表格文件
OTU/ASV/基因注释文件 (从此处注释文件中选择分析所用分类水平,如:Phylum,Genus)

小工具结果:
Lefse检验结果.csv

LDA_score_range.csv
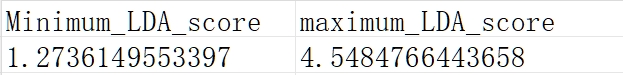
LDA值分布柱状图.pdf
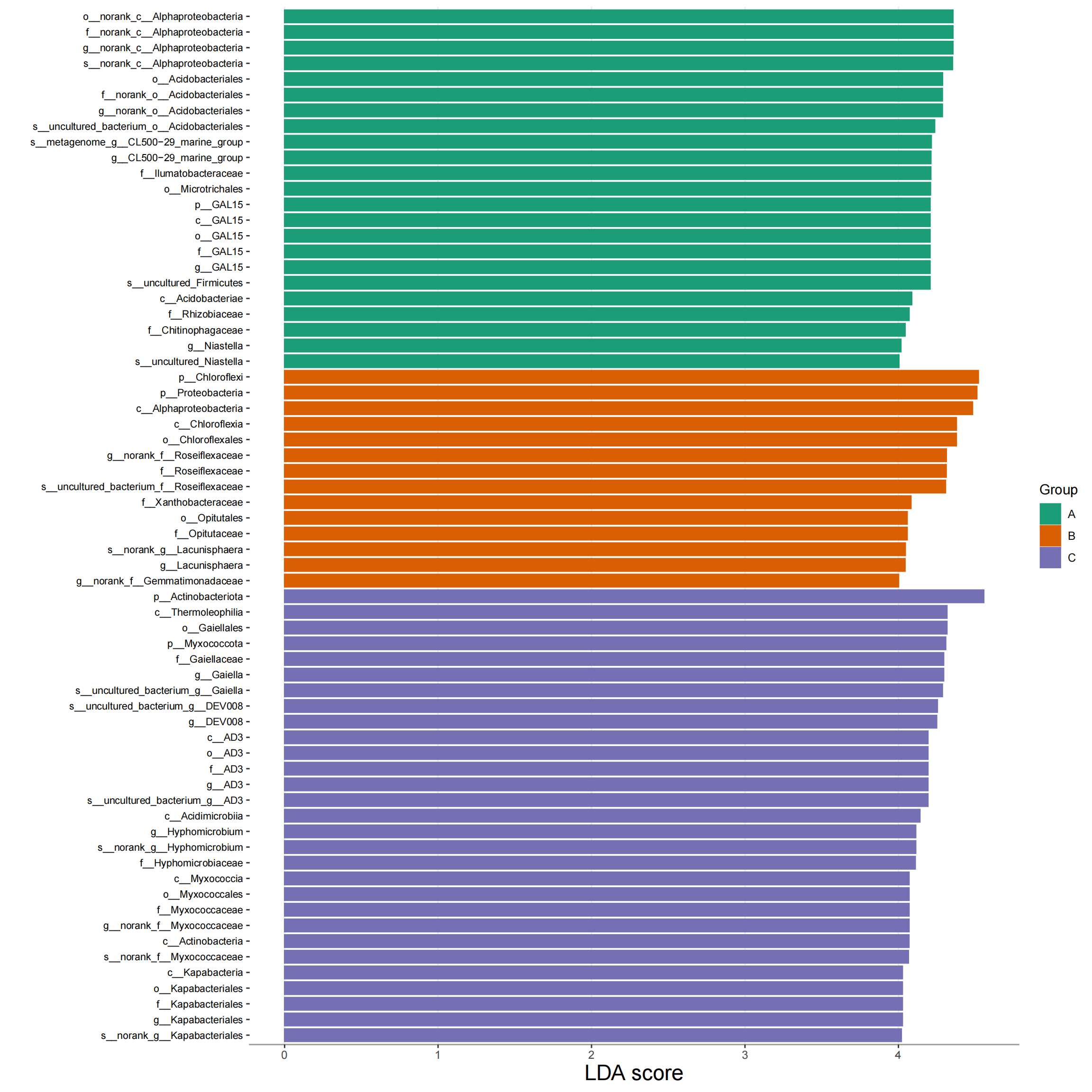
物种进化分支图.pdf

使用场景
- 比较不同组别之间微生物组成差异
- 筛选出具有统计学差异的生物标识(Biomarker )
当前版本为1.0版本,上架时间为:2025-05-12