- 结果文件
- 使用指南
- 版本记录
- 联系客服
本工具用于识别质粒的多位点基因型(MLST)、双位点基因型(double-locus sequence typing,DLST)以及复制子基因型(replicon sequence typing,RST)。其参考序列数据来自PubMLST(BIGSdb数据库的成员)的pMLST数据库,涵盖IncA/C、IncF、IncHI1、IncHI2、IncI1,和IncN这六种不相容性(incompatibility)群。与PlasmidFinder相同,pMLST接受FASTQ格式reads以及FASTA格式组装序列输入。
pMLST 支持以下质粒分型方案:
- 1.
incac- IncA/C 不相容群 - 2.
incf- IncF 不相容群 - 3.
inchi1- IncHI1 不相容群 - 4.
inchi2- IncHI2 不相容群 - 5.
inci1- IncI1 不相容群 - 6.
incn- IncN 不相容群 - 7.
pbssb1-family- PBS1 噬菌体单链结合蛋白家族 - 8.
shigella- 志贺氏菌质粒
如何选择合适的方案
选择合适的方案取决于您要分析的质粒类型:
- 1.临床分离株:通常使用
incf(IncF 质粒常见于临床环境) - 2.多重耐药质粒:考虑
incac或inchi1/2 - 3.志贺氏菌:使用
shigella - 4.如果不确定:可以尝试运行多个方案或使用质粒查找工具(如 PlasmidFinder)先确定质粒类型
输入文件:
FASTA格式组装序列输入
小工具结果:
results.txt
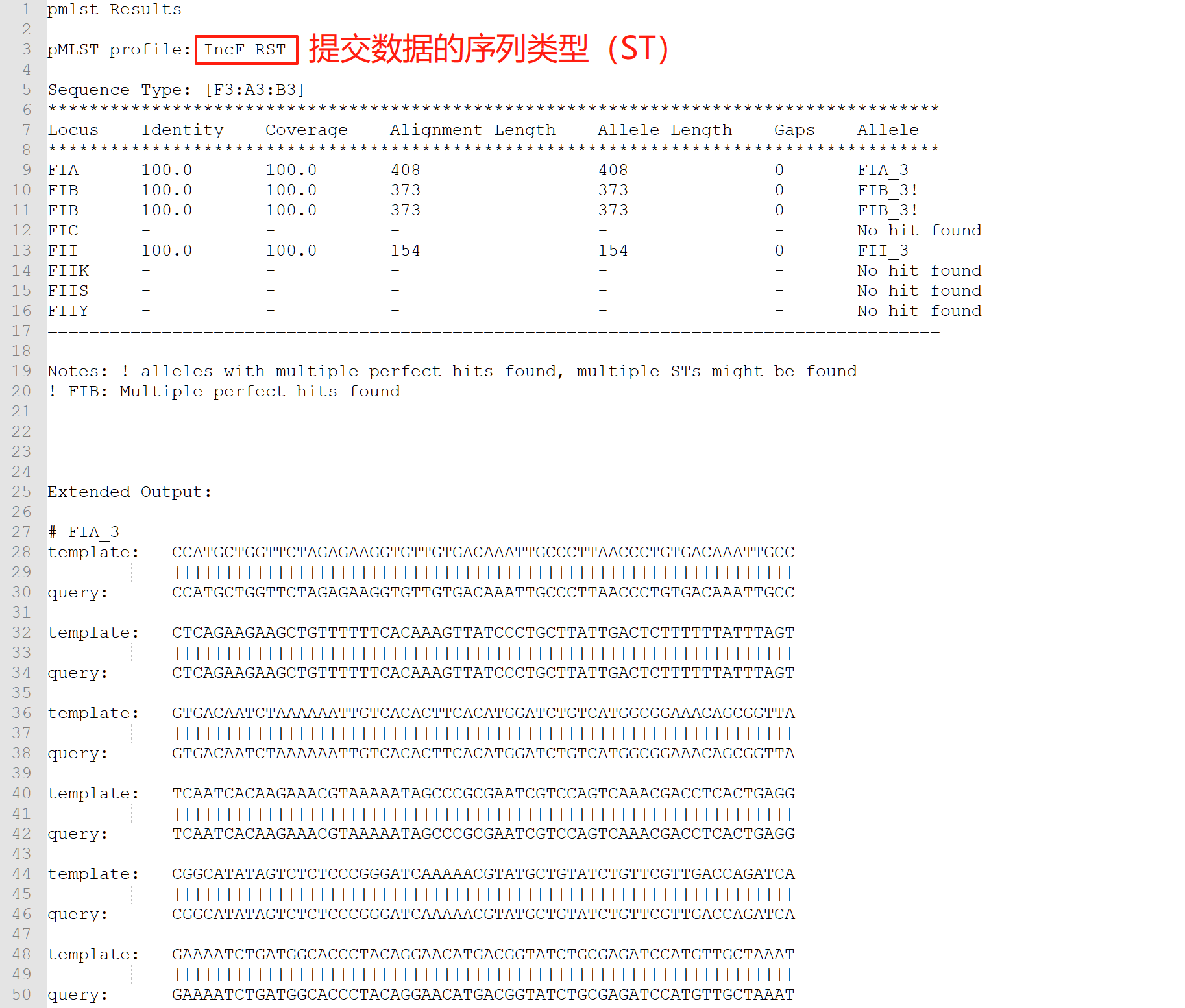
软件版本:2.0.1-5-g5618093
参考文献:
Lund O , Villa L , Aarestrup F M , et al. PlasmidFinder and pMLST: in silico detection and typing of plasmids.[J]. antimicrobial agents & chemotherapy.
当前版本为1.0版本,上架时间为:2025-08-22