- 结果文件
- 使用指南
- 版本记录
- 联系客服
本工具用于根据全基因组序列组装数据对铜绿假单胞菌(Pseudomonas aeruginosa)分离株进行快速可靠的计算机血清分型。
输入文件:
包含基因组组装结果(fasta格式)的文件夹路径
小工具结果:
serotyping.txt:预测血清群的结果摘要
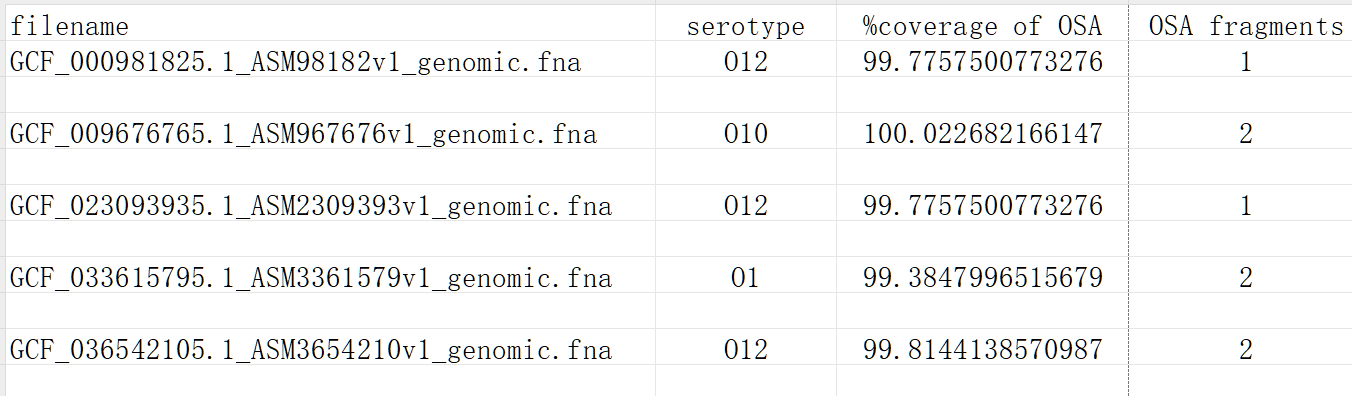
filename :输入的待分析基因组文件的名称;serotype :血清型,PAst分析得出的最终结论,即该铜绿假单胞菌菌株属于哪个O血清型;%coverage of OSA: O抗原基因簇覆盖率,表示被测基因组的序列与PAst数据库中该血清型标准O抗原基因簇(O-antigen gene cluster)的匹配覆盖程度,以百分比形式显示,值越高越好,通常>90%或95%就认为覆盖率非常好,结果非常可靠,如果这个值非常低(例如<70%),则可能意味着分型结果不可靠,需要进一步检查;OSA fragments :O抗原基因簇碎片数,表示被测基因组的O抗原基因簇是由多少条独立的DNA序列片段(Contigs)组装而成的,值越低越好,理想状态是 1,意味着完整的O抗原基因簇被完整地组装在一条连续的序列中,没有任何中断,这表明组装质量极高,结果最可靠,2 表示该基因簇由2条片段组成,组装质量很好,如果这个数值很高(例如>5),说明基因组组装比较碎片化,O抗原基因簇可能被拆得很散。这可能会影响%coverage的计算和分型结果的准确性,需要谨慎对待。
BLASTGCF_*.fna:每个输入基因组的 BLAST 报告
O*-OSA_*.fna:从每个输入基因组中提取的 OSA 簇的 Multifasta 文件
软件版本:Pseudomonas aeruginosa serotyper (PAst) v3
当前版本为1.0版本,上架时间为:2025-07-25