- 结果文件
- 使用指南
- 版本记录
- 联系客服
使用了 MMseqs2 软件进行生物序列的分类注释和结果整理。
MMseqs2 是一个高效的序列比对和数据库搜索工具,广泛应用于大规模基因组数据的处理和分析。
GTDB(Genome Taxonomy Database)是一个专注于基因组分类学的数据库,旨在提供高质量的细菌、古细菌、叶绿体和线粒体的分类信息。通过使用全基因组信息,GTDB提供了多级别的分类,包括域、界、纲、目、科、属等级别。其系统发育分类系统使得研究人员能够深入了解生物的演化和功能。GTDB主要以细菌中普遍存在的120个单拷贝蛋白质(bac120)和古菌中53个(arc53,从R07-RS207开始)标记蛋白为基础,在对多分组类别消歧后,根据相对演化散度标准化和分级,得到基因组分类结果。
示例文件:
待注释contig文件,包含需要进行注释的contig,后缀名为*.fasta、*.fa、*.fas、*.fna、*.fnn等。

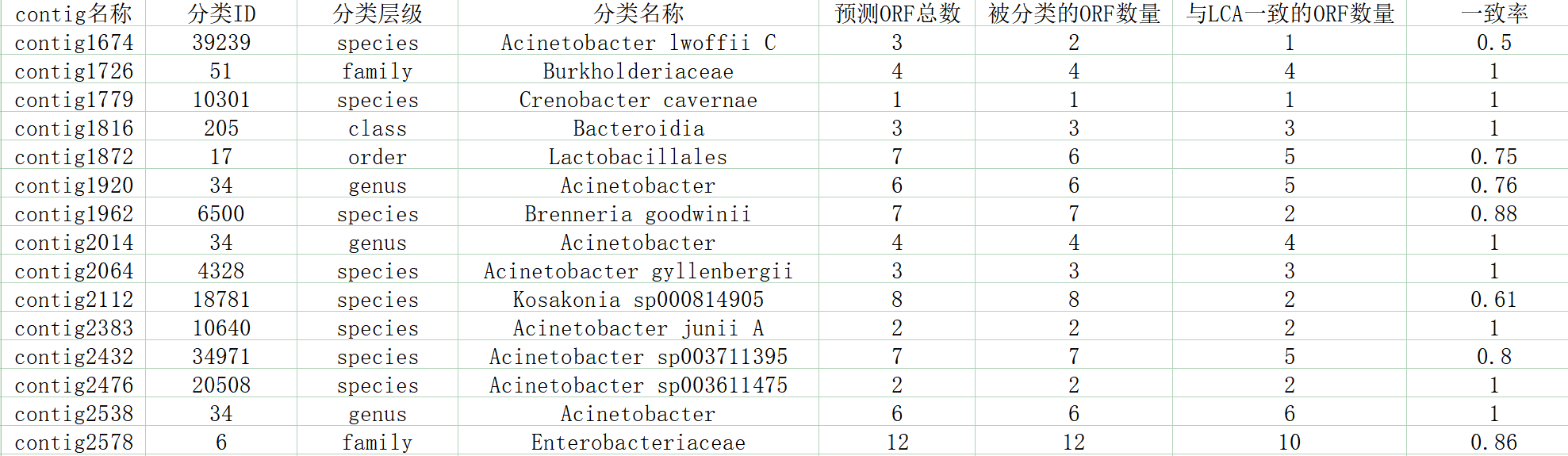
小工具结果:
taxonomyResult.tsv
参考文献:
- Parks, D.H., et al. (2021). "GTDB: an ongoing census of bacterial and archaeal diversity through a phylogenetically consistent, rank normalized and complete genome-based taxonomy.
" Nucleic Acids Research, https://doi.org/10.1093/nar/gkab776. - Steinegger M and Soeding J. MMseqs2 enables sensitive protein sequence searching for the analysis of massive data sets. Nature Biotechnology, doi: 10.1038/nbt.3988 (2017).
当前版本为1.0版本,上架时间为:2025-07-10