- 结果文件
- 使用指南
- 版本记录
- 联系客服
此工具用来计算多序列比对文件的多态性(pi),输入文件可以是核酸的多序列比对文件,也可以是蛋白的多序列比对文件。支持滑动窗口分析,输出整个区域的总体pi值,以及每个滑动窗口区域的 pi和变异位点数。
多态性(pi)是群体遗传学中用来衡量一个群体内核苷酸水平的遗传多样性的一个指标。它反映了群体中任意两个个体在基因组某一位点上平均的碱基差异程度。
多态性越高,表示群体内部遗传多样性高,说明突变频繁、遗传资源丰富,可能适应力强。反之,多态性低可能说明群体经历了瓶颈效应、强选择压力,遗传多样性低。
示例
输入数据:4条核酸序列的多序列比对文件

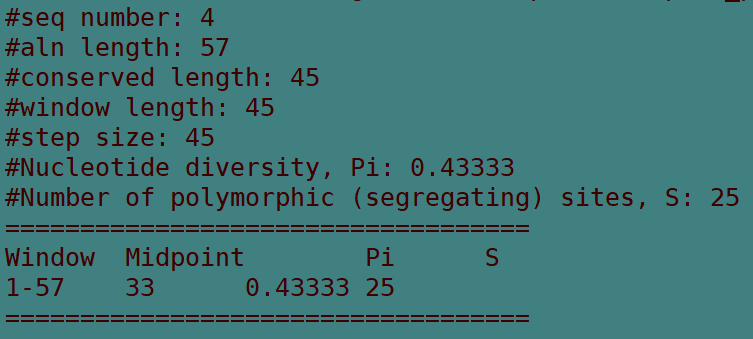
输出结果:
seq number:输入的序列数量(样本数)
aln length:多序列比对的长度
conserved length:剔除indel后保留的碱基位点数,即用于pi计算的位点数
window length:滑动窗口长度,默认为序列总长,即conserved length
step size:滑动步长,默认为序列总长,即conserved length
Nucleotide diversity, Pi:全区域平均核苷酸多态性(pi 值),反映样本整体遗传多样性
Number of polymorphic sites, S:全区域的多态位点总数
Window:当前滑窗范围
Midpoint:当前窗口的中点位点,方便绘图或定位
Pi:当前窗口区域内的核苷酸多态性 π 值,越高表示该区域多样性越高
S:当前窗口内的多态位点数(segregating sites)
暂无内容