- 结果文件
- 使用指南
- 版本记录
- 联系客服
PointFinder 是一个用于检测细菌基因组中已知点突变耐药位点(point mutations related to antimicrobial resistance, AMR)的软件工具,尤其适用于在大肠杆菌、沙门氏菌、志贺菌、弯曲杆菌等重要病原体中进行已知耐药突变筛查。
分析原理:通过 BLAST 或 BWA 将输入序列与 PointFinder 数据库中对应物种的参考基因序列进行比对,识别在参考基因位置上发生的碱基变异,判断是否为已知的抗性突变位点。输出突变列表和对应的抗性药物解释
可分析的物种包括campylobacter(弯曲杆菌)、enterococcus_faecalis(粪肠球菌)、enterococcus_faecium(屎肠球菌)、escherichia_coli(大肠杆菌)、helicobacter_pylori(幽门螺杆菌)、klebsiella(克雷伯氏菌属细菌)、mycobacterium_tuberculosis(结核分枝杆菌)、neisseria_gonorrhoeae(淋病奈瑟菌)、plasmodium_falciparum(恶性疟原虫)、salmonella(沙门菌)、staphylococcus_aureus(金黄色葡萄球菌)。
输出结果:
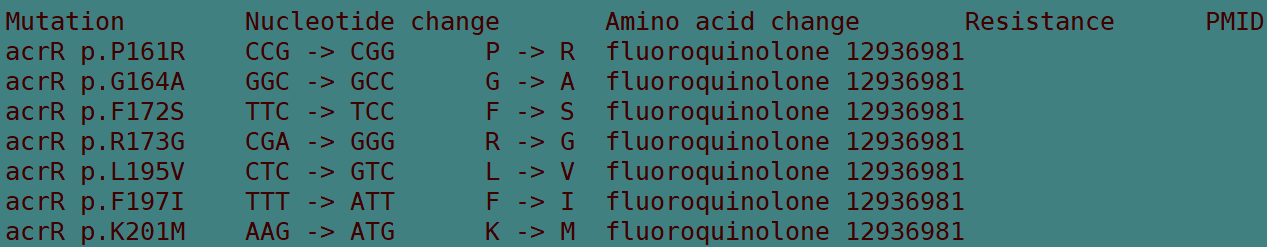
1、*_blastn_results.tsv 突变详情
共5列
第一列,Mutation,突变发生的基因,以及发生位点,如acrR p.P161R表示acrR基因第161位的氨基酸由P突变为R;
第二列,Nucleotide change。核酸序列发生的变异,如CCG -> CGG表示发生了CCG到CGG的突变;
第三列,Amino acid change,氨基酸变异,如P -> R表示氨基酸由P突变为R;
第四列,Resistance,即该基因对应的抗性;
第五列,PMID,对应的文献编号;
2、*_blastn_prediction.txt 抗性基因分类统计表
第一列是样本名称,后面各列是抗生素类型,数字表示样本中包含对应抗性基因的数量。

此版本为1.0,上架时间为20250616