- 结果文件
- 使用指南
- 版本记录
- 联系客服
ABRicate是一款用于快速分析微生物基因组数据的软件,可以基于细菌基因组组装结果进行分析,软件自带多个数据库,可以帮助我们更轻松的检测抗生素耐药基因和毒力因子等。
支持以下数据库:
| NCBI | 基于NCBI的耐药数据库 |
| CARD | 基于CARD的耐药数据库 |
| VFDB | 基于vfdb毒力基因数据库 |
| ARG-ANNOT | 基于argannot耐药数据库 |
| Resfinder | 基于resfinder耐药数据库 |
| MEGARES | 基于megares耐药数据库 |
| EcOH | 用于大肠杆菌O/H抗原分析 |
| Ecoli_VF | 基于大肠杆菌毒力基因 |
| PlasmidFinder | 基于plasmidfinder用于质粒扫描 |
输入文件:
Fasta格式的基因组预组装结果文件

小工具结果:
*_out.txt:根据选择的数据库输出对应的注释结果,如:ncbi_out.txt
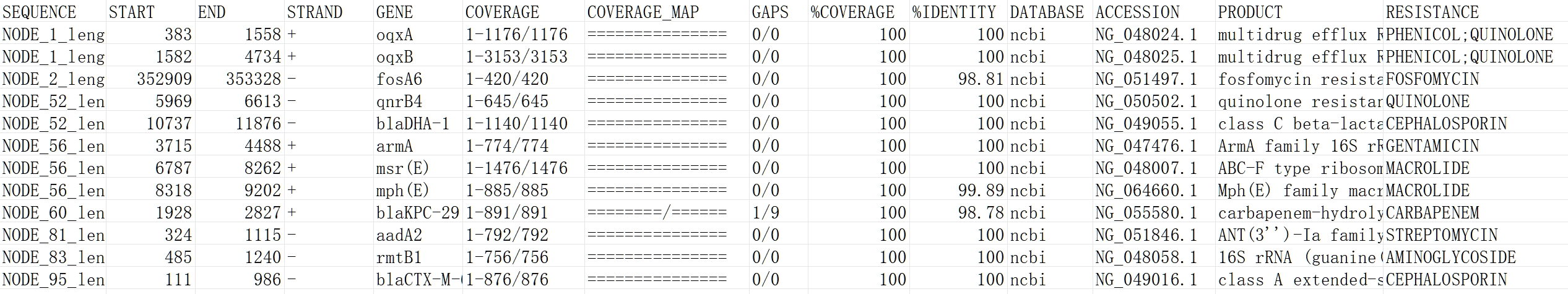
SEQUENCE:序列名称;START:匹配区域在序列上的起始位置;END:匹配区域在序列上的终止位置;STRAND:匹配区域所在的链,"+"表示正链,"-"表示负链;GENE:基因/质粒分型基因;COVERAGE:匹配的碱基范围及总长度,格式为“起止-终止/总长”,如1-251/251表示全长覆盖;COVERAGE_MAP:匹配覆盖的可视化表示,"="表示覆盖,"/"表示缺口;GAPS:匹配中的缺口数,格式为“实际缺口数/允许缺口数”;%COVERAGE:匹配区域占参考基因的百分比,100表示全长覆盖;%IDENTITY:匹配区域与参考基因的相似性百分比;DATABASE:匹配所用的数据库名称,如ncbi(基于NCBI的耐药数据库),plasmidfinder(质粒分型数据库);ACCESSION:参考基因在数据库中的登录号(Accession Number);PRODUCT:匹配到的基因或产物的详细名称;RESISTANCE:相关的耐药性信息
软件版本:abricate 1.0.1
https://github.com/tseemann/abricate
当前版本为1.0版本,上架时间为:2025-05-19