- 结果文件
- 使用指南
- 版本记录
- 联系客服
识别和分析宏基因组数据中的噬菌体,小工具基于PhaBOX开发,集成了工具(PhaMer、PhaTYP、PhaGCN 和 CHERRY),分别用于噬菌体鉴定、生活方式预测、分类学分类和宿主预测
支持以下参数:
end_to_end:全流程一键执行整合以下5个核心模块(phamer → phagcn → phatyp → cherry → phavip),自动完成从病毒鉴定到功能注释的全流程分析;
phamer:病毒序列鉴定通过比对数据库(如Caudoviricetes类病毒)识别输入序列中的病毒Contig,过滤宿主污染;
phagcn:分类学分级,基于图卷积神经网络(GCN)与Virus Taxonomic Database,预测病毒的科/属级分类地位;
phatyp:溶原/裂解生活方式预测,通过机器学习模型(整合基因含量、序列特征)判断病毒是:✅ 温和性噬菌体(可整合宿主基因组) ✅ 裂解性噬菌体(直接裂解宿主);
cherry:宿主预测,基于CRISPR spacer匹配、同源基因及序列相似性,预测病毒可能侵染的宿主物种(如细菌/古菌);
phavip:病毒蛋白功能注释,比对PHROG、VFDB等数据库,注释病毒蛋白的功能域(如衣壳蛋白、整合酶)、毒力因子及保守基因家族;
contamination:污染检测,识别样本中的外源污染(如宿主DNA、质粒)或前噬菌体(Provirus),确保数据纯净性;
votu:病毒操作分类单元聚类,基于ANI(核酸相似性)或AAI(氨基酸相似性)将病毒基因组聚类为vOTUs,用于界定物种边界(通常≥95% ANI为同一vOTU);
tree:系统发育树构建,利用噬菌体保守标记基因(如Terminase大亚基)构建多序列比对,生成系统发育树,可视化病毒间的进化关系。
示例文件:
待注释contig文件,后缀名为*.fasta、*.fa、*.fas、*.fna、*.fnn等,提交文件后设置长度截止阈值,要求>=3000bp,如:3000

小工具结果:
final_prediction_summary.tsv:通过合并所有子程序的输出而生成
phavip_prediction.tsv
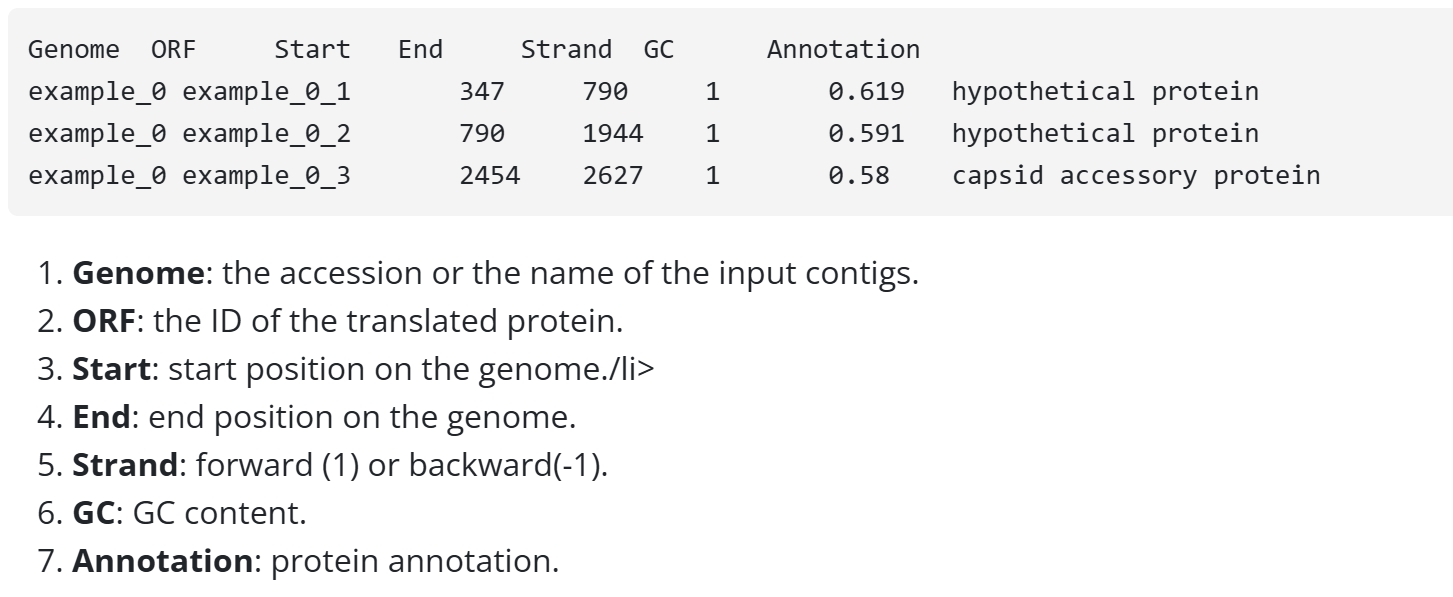
phamer_prediction.tsv

cherry_prediction.tsv

phagcn_prediction.tsv
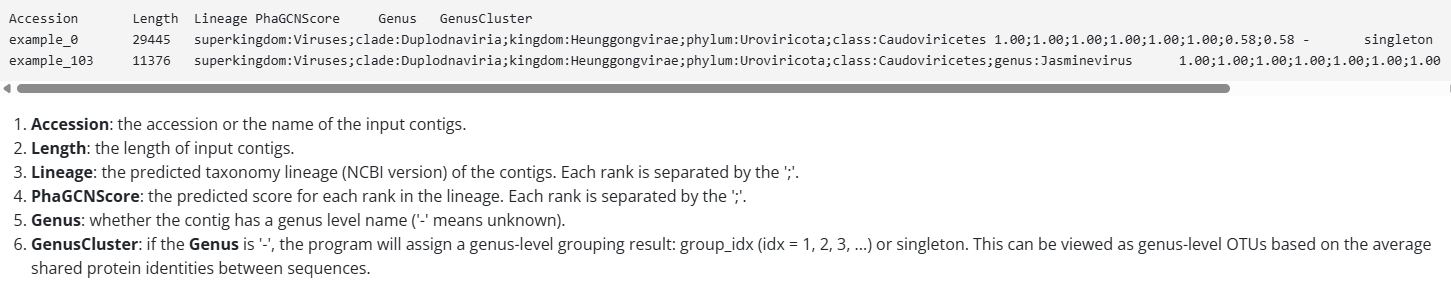
phatyp_prediction.tsv

参考文献:
1. Jiayu Shang, Cheng Peng, Herui Liao, Xubo Tang, Yanni Sun, PhaBOX: a web server for identifying and characterizing phage contigs in metagenomic data, Bioinformatics Advances, Volume 3, Issue 1, 2023, vbad101, https://doi.org/10.1093/bioadv/vbad101
2. Jiayu Shang, Xubo Tang, Ruocheng Guo, Yanni Sun, Accurate identification of bacteriophages from metagenomic data using Transformer, Briefings in Bioinformatics, 2022;, bbac258, https://doi.org/10.1093/bib/bbac258
3. Jiayu Shang, Jingzhe Jiang, Yanni Sun, Bacteriophage classification for assembled contigs using graph convolutional network, Bioinformatics, Volume 37, Issue Supplement_1, July 2021, Pages i25–i33, https://doi.org/10.1093/bioinformatics/btab293
4. Jiayu Shang, Yanni Sun, CHERRY: a Computational metHod for accuratE pRediction of virus–pRokarYotic interactions using a graph encoder–decoder model, Briefings in Bioinformatics, 2022;, bbac182, https://doi.org/10.1093/bib/bbac182
5. Jiayu Shang, Xubo Tang, Yanni Sun, PhaTYP: predicting the lifestyle for bacteriophages using BERT, Briefings in Bioinformatics, 2022;, bbac487, https://doi.org/10.1093/bib/bbac487
当前版本为1.0版本,上架时间为:2025-07-06