- 结果文件
- 使用指南
- 版本记录
- 联系客服
本工具用于从宏基因组数据中挖掘抗菌肽(Antimicrobial Peptides,AMPs)。输入文件为宏基因组的组装结果Fasta格式文件,或预测的蛋白序列文件。
示例文件:
宏基因组的组装结果,选择:contigs
预测的蛋白序列文件,选择:peptides
小工具结果:
macrel.out.prediction
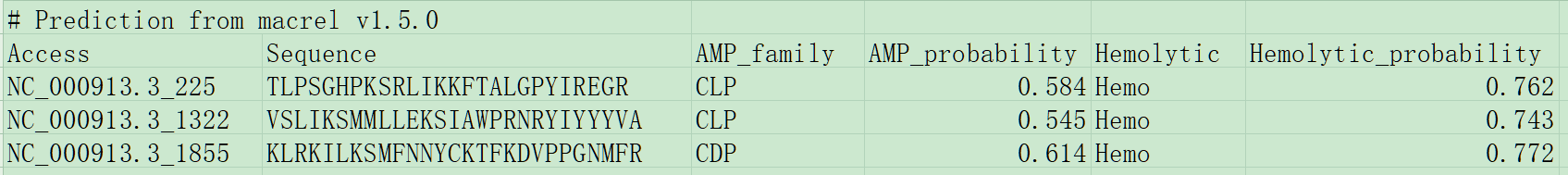
Access:序列标识符;Sequence:预测的氨基酸序列;AMP_family:AMP 家族分类,A/C:阴离子/阳离子肽, D/L:含半胱氨酸/线性肽, 后缀 P表示肽段(例:阳离子含半胱氨酸肽 → CDP);AMP_probability:该序列为抗菌肽的概率(0-1);Hemolytic:溶血性分类,Hemo(溶血性)/NonHemo(非溶血性);Hemolytic_probability:溶血活性的概率(0-1)。
macrel.out.all_orfs.faa:全部预测的开放阅读框(ORFs)
macrel.out.smorfs.faa:预测的小型 ORFs(smORFs),长度范围限定为 10-100 个氨基酸。
macrel.out.percontigs:每个 contig 的统计摘要
列名 | 说明 |
|---|---|
| Contig 标识符(与输入 FASTA 一致; |
| Contig 长度(单位:bp) |
| 预测的 ORF 总数 |
| 预测的小型 ORF(smORF)数量 |
| 预测为抗菌肽(AMP)的序列数量 |
软件版本:
macrel v1.5.0
参考文献:
Santos-Júnior CD, Pan S, Zhao X, Coelho LP. 2020. Macrel: antimicrobial peptide screening in genomes and metagenomes. PeerJ 8:e10555. DOI: 10.7717/peerj.10555
当前版本为1.0版本,上架时间为:2025-06-13